| DLD |
|---|
 |
| Struktur sedia ada |
|---|
| PDB | Pencarian ortolog: PDBe RCSB |
|---|
| Senarai kod PDB |
|---|
3RNM, 1ZMC, 1ZMD, 1ZY8, 2F5Z |
|
|
| Pengecam |
|---|
| Alias | DLD, DLDD, DLDH, E3, GCSL, LAD, PHE3, dihydrolipoamide dehydrogenase, Dihydrolipoamide dehydrogenase, OGDC-E3 |
|---|
| Pengecam-pengecam luaran | OMIM: 238331 MGI: 107450 HomoloGene: 84 GeneCards: DLD |
|---|
| Kedudukan gen (Manusia) |
|---|
 | | Kr. | Kromosom 7[1] |
|---|
| | Jalur | 7q31.1 | Mula | 107,891,162 bp[1] |
|---|
| Tamat | 107,931,730 bp[1] |
|---|
|
| Corak ekspresi RNA |
|---|
| Bgee | | Human | Mouse (ortholog) |
|---|
| Top expressed in | - right ventricle
- buah lengan
- Skeletal muscle tissue of biceps brachii
- Diafragma
- Skeletal muscle tissue of rectus abdominis
- vastus lateralis muscle
- body of tongue
- mucosa of sigmoid colon
- mukosa jejunum
- rektum
|
| | Top expressed in | - saccule
- cardiac muscle tissue of left ventricle
- otic placode
- tail of embryo
- extraocular muscle
- intercostal muscle
- soleus muscle
- extensor digitorum longus muscle
- interventricular septum
- plantaris muscle
|
| | More reference expression data |
|
|---|
| BioGPS | |
|---|
|
| Ontologi gen |
|---|
| Fungsi molekul | - flavin adenine dinucleotide binding
- NAD binding
- dihydrolipoyl dehydrogenase activity
- oxidoreductase activity
- pyruvate dehydrogenase (NAD+) activity
- lipoamide binding
- oxidoreductase activity, acting on a sulfur group of donors, NAD(P) as acceptor
- electron transfer activity
- Pengikatan protein plasma
| | Komponen sel | - Kompleks oksoglutarat dehidrogenase
- myelin sheath
- Kompleks piruvat dehidrogenase
- Silium
- acrosomal matrix
- nucleoplasm
- mitochondrial matrix
- Mitokondrion
- acrosomal vesicle
- cytoplasmic vesicle
- motile cilium
- cell projection
- nukleus sel
| | Proses biologi | - mitochondrial acetyl-CoA biosynthetic process from pyruvate
- regulation of membrane potential
- penuaan
- branched-chain amino acid catabolic process
- Kitar Krebs
- 2-oxoglutarate metabolic process
- lysine catabolic process
- regulation of acetyl-CoA biosynthetic process from pyruvate
- cell redox homeostasis
- Proteolisis
- lipoate metabolic process
- dihydrolipoamide metabolic process
- gastrulation
- pyruvate metabolic process
- sperm capacitation
- acetyl-CoA biosynthetic process from pyruvate
- mitochondrial electron transport, NADH to ubiquinone
- cellular nitrogen compound metabolic process
- histone succinylation
| | Sumber:Amigo / QuickGO |
|
| Ortolog |
|---|
| Spesies | Manusia | Mencit |
|---|
| Entrez | | |
|---|
| Ensembl | | |
|---|
| UniProt | | |
|---|
| RefSeq (mRNA) | |
|---|
NM_001289752
NM_000108
NM_001289750
NM_001289751 |
| |
|---|
| RefSeq (protein) | |
|---|
NP_000099
NP_001276679
NP_001276680
NP_001276681 |
| |
|---|
| Kedudukan (UCSC) | Chr 7: 107.89 – 107.93 Mb | tiada data |
|---|
| Carian PubMed | [2] | [3] |
|---|
|
| Wikidata |
| Papar/Sunting data manusia | Papar/Sunting data mencit |
|
Dihidrolipoamida dehidrogenase (DLD), juga dikenali sebagai dihidrolipoil dehidrogenase mitokondrion, ialah enzim yang dikodkan oleh gen DLD dalam manusia.[4][5][6][7] DLD ialah enzim flavoprotein yang mengoksidakan dihidrolipoamida kepada lipoamida. Dihidrolipoamida dehidrogenase (DLD) ialah enzim mitokondria yang memainkan peranan penting dalam metabolisme tenaga dalam eukariot. Enzim ini diperlukan bagi tindak balas lengkap sekurang-kurangnya lima kompleks pelbagai enzim yang berbeza.[8] Selain itu, DLD ialah flavoenzim oksidoreduktase yang mengandungi jambatan disulfida reaktif dan kofaktor FAD yang terlibat secara langsung dalam pemangkinan. Enzim bersekutu ke dalam homodimer terikat ketat yang diperlukan bagi aktiviti enzimatiknya.[9]
-
Lipoamida
-
Dihidrolipoamida
Struktur
Protein yang dikodkan oleh gen DLD datang bersama dengan protein lain untuk membentuk dimer dalam laluan metabolik pusat. Beberapa asid amino dalam poket pemangkin telah dikenal pasti sebagai penting dalam fungsi DLD, termasuk R281 dan N473.[10][11] Walaupun lipatan keseluruhan enzim manusia adalah serupa dengan yis, struktur manusia berbeza kerana ia mempunyai dua gelung yang memanjang dari struktur protein umum ke dalam tapak pengikatan FAD ketika terikat molekul NAD+ yang diperlukan untuk pemangkinan tidak dekat dengan bahagian FAD. Walau bagaimanapun, apabila NADH diikat pula, ia disusun terus di atas struktur pusat FAD. Struktur hE3 semasa menunjukkan secara langsung bahawa mutasi penyebab penyakit berlaku di tiga lokasi dalam enzim manusia: antara muka dimer, tapak aktif dan tapak pengikat FAD dan NAD(+).[12]
Fungsi
Homodimer DLD berfungsi sebagai komponen E3 bagi kompleks dehidrogenase piruvat, α-ketoglutarat, α-adipat dan asid amino rantai bercabang serta sistem pembelahan glisina, semuanya dalam matriks mitokondria. Dalam kompleks ini, DLD menukarkan asid dihidrolipoik dan NAD+ kepada asid lipoik dan NADH.[13] DLD juga mempunyai aktiviti diaforase, yakni dapat memangkinkan pengoksidaan NADH kepada NAD+ dengan menggunakan penerima elektron yang berbeza seperti O2, besi ferik pendam, nitrik oksida dan ubikuinon.[8] DLD dianggap mempunyai peranan propengoksidaan dengan menurunkan oksigen menjadi superoksida atau besi ferik kepada ferus, yang kemudian memangkinkan pengeluaran radikal hidroksil.[14][15] Aktiviti diaforase DLD mungkin mempunyai peranan antioksidan melalui keupayaannya untuk mengais oksida nitrik, dan menurunkan ubikuinon kepada ubikuinol.[16][17][18] Gen dihirolipamida dehidrogenase diketahui mempunyai pelbagai variasi penyambatan.
Fungsi sampingan
Mutasi DLD tertentu secara serentak boleh menyebabkan kehilangan aktiviti metabolisme utama dan keuntungan aktiviti proteolisis aktiviti sampingan. Aktiviti proteolisis sampingan DLD didedahkan oleh keadaan yang mengganggu kestabilan homodimer DLD dan mengurangkan aktiviti DLD-nya.[8] Pengasidan matriks mitokondria akibat kecederaan iskemia-reperfusi boleh mengganggu struktur kuaterner DLD, membawa kepada penurunan aktiviti dehidrogenase dan peningkatan aktiviti diaforase.[19] Aktiviti proteolisis sampingan DLD juga boleh timbul di bawah keadaan patologi. Aktiviti proteolisis boleh merumitkan lagi pengurangan metabolisme tenaga dan peningkatan kerosakan oksidatif akibat penurunan aktiviti DLD dan peningkatan aktiviti diaforase masing-masing.[18] Dengan fungsi proteolisisnya, DLD membuang domain yang penting dari segi fungsi dari terminal N frataksin, protein mitokondria yang terlibat dalam metabolisme besi dan perlindungan antioksidan.[20][21]
Kepentingan klinikal
Pada manusia, mutasi dalam DLD dikaitkan dengan gangguan bayi yang teruk dengan kegagalan berkembang, hipotonia dan asidosis metabolisme.[22] Kekurangan DLD menunjukkan dirinya dalam tahap kebolehubahan yang besar, yang telah dikaitkan dengan pelbagai kesan mutasi DLD yang berbeza terhadap kestabilan protein dan keupayaannya untuk dimerisasi atau berinteraksi dengan komponen lain dari tiga kompleks dehidrogenase asid α-ketoa.[22] Dengan fungsi proteolitiknya, DLD menyebabkan kekurangan frataksin, yang membawa kepada penyakit neurodegeneratif dan jantung, ataksia Friedreich.[23]
Kawal atur enzim
Protein ini mungkin menggunakan model morfein dalam kawal atur alosterik.[24]
Rujukan
- ^ a b c GRCh38: Ensembl release 89: ENSG00000091140 - Ensembl, May 2017
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Mouse PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Entrez Gene: dihydrolipoamide dehydrogenase".
- ^ "Isolation and sequence determination of cDNA clones for porcine and human lipoamide dehydrogenase. Homology to other disulfide oxidoreductases". The Journal of Biological Chemistry. 262 (36): 17313–8. December 1987. doi:10.1016/S0021-9258(18)45379-3. PMID 3693355.
- ^ "Cloning and cDNA sequence of the dihydrolipoamide dehydrogenase component human alpha-ketoacid dehydrogenase complexes". Proceedings of the National Academy of Sciences of the United States of America. 85 (5): 1422–6. March 1988. Bibcode:1988PNAS...85.1422P. doi:10.1073/pnas.85.5.1422. PMC 279783. PMID 3278312. Unknown parameter
|displayauthors= ignored (bantuan) - ^ "Localization of the human dihydrolipoamide dehydrogenase gene (DLD) to 7q31----q32". Cytogenetics and Cell Genetics. 56 (3–4): 176–7. 1991. doi:10.1159/000133081. PMID 2055113.
|hdl-access= requires |hdl= (bantuan) - ^ a b c "Cryptic proteolytic activity of dihydrolipoamide dehydrogenase". Proceedings of the National Academy of Sciences of the United States of America. 104 (15): 6158–63. April 2007. Bibcode:2007PNAS..104.6158B. doi:10.1073/pnas.0610618104. PMC 1851069. PMID 17404228.
- ^ "How dihydrolipoamide dehydrogenase-binding protein binds dihydrolipoamide dehydrogenase in the human pyruvate dehydrogenase complex". The Journal of Biological Chemistry. 281 (1): 648–55. January 2006. doi:10.1074/jbc.M507850200. PMID 16263718.
- ^ "Asparagine-473 residue is important to the efficient function of human dihydrolipoamide dehydrogenase". Journal of Biochemistry and Molecular Biology. 38 (2): 248–52. March 2005. doi:10.5483/bmbrep.2005.38.2.248. PMID 15826505.
- ^ "The role of amino acids T148 and R281 in human dihydrolipoamide dehydrogenase". Journal of Biomedical Science. 15 (1): 37–46. January 2008. doi:10.1007/s11373-007-9208-9. PMID 17960497. Unknown parameter
|displayauthors= ignored (bantuan) - ^ "Crystal structure of human dihydrolipoamide dehydrogenase: NAD+/NADH binding and the structural basis of disease-causing mutations". Journal of Molecular Biology. 350 (3): 543–52. July 2005. doi:10.1016/j.jmb.2005.05.014. PMID 15946682.
- ^ "Dihydrolipoamide dehydrogenase: functional similarities and divergent evolution of the pyridine nucleotide-disulfide oxidoreductases". Archives of Biochemistry and Biophysics. 268 (2): 409–25. February 1989. doi:10.1016/0003-9861(89)90309-3. PMID 2643922.
- ^ "Reduction of Fe(III) ions complexed to physiological ligands by lipoyl dehydrogenase and other flavoenzymes in vitro: implications for an enzymatic reduction of Fe(III) ions of the labile iron pool". The Journal of Biological Chemistry. 278 (47): 46403–13. November 2003. doi:10.1074/jbc.M305291200. PMID 12963736. Unknown parameter
|displayauthors= ignored (bantuan) - ^ "Proteomic identification of dihydrolipoamide dehydrogenase as a target of autoantibodies in patients with endometrial cancer". Anticancer Research. 34 (9): 5021–7. September 2014. PMID 25202086. Unknown parameter
|displayauthors= ignored (bantuan) - ^ "Dihydrolipoamide dehydrogenase from porcine heart catalyzes NADH-dependent scavenging of nitric oxide". FEBS Letters. 568 (1–3): 146–50. June 2004. doi:10.1016/j.febslet.2004.05.024. PMID 15196936.
- ^ "Ubiquinone is reduced by lipoamide dehydrogenase and this reaction is potently stimulated by zinc". FEBS Letters. 448 (1): 190–2. April 1999. doi:10.1016/s0014-5793(99)00363-4. PMID 10217438.
- ^ a b "Reduction of ubiquinone by lipoamide dehydrogenase. An antioxidant regenerating pathway". European Journal of Biochemistry. 268 (5): 1486–90. March 2001. doi:10.1046/j.1432-1327.2001.02013.x. PMID 11231302.
- ^ "pH-dependent substrate preference of pig heart lipoamide dehydrogenase varies with oligomeric state: response to mitochondrial matrix acidification". The Journal of Biological Chemistry. 280 (16): 16106–14. April 2005. doi:10.1074/jbc.M414285200. PMID 15710613.
- ^ "Chelatases: distort to select?". Trends in Biochemical Sciences. 31 (3): 135–42. March 2006. doi:10.1016/j.tibs.2006.01.001. PMC 2997100. PMID 16469498.
- ^ "Assembly of human frataxin is a mechanism for detoxifying redox-active iron". Biochemistry. 44 (2): 537–45. January 2005. doi:10.1021/bi048459j. PMID 15641778. Unknown parameter
|displayauthors= ignored (bantuan) - ^ a b Quinonez SC, Thoene JG (9 July 2020). "Dihydrolipoamide Dehydrogenase Deficiency". Dalam Adam MP, Ardinger HH, Pagon RA, Wallace SE, Bean LJ, Mirzaa G, Amemiya A (penyunting). GeneReviews. University of Washington, Seattle. PMID 25032271.
- ^ "Human dihydrolipoamide dehydrogenase (E3) deficiency: Novel insights into the structural basis and molecular pathomechanism" (PDF). Neurochemistry International. 117: 5–14. July 2018. doi:10.1016/j.neuint.2017.05.018. PMID 28579060.
- ^ "Dynamic dissociating homo-oligomers and the control of protein function". Archives of Biochemistry and Biophysics. 519 (2): 131–43. March 2012. doi:10.1016/j.abb.2011.11.020. PMC 3298769. PMID 22182754.
Pautan luar
|
|---|
| 1.8.1: NAD atau NADP | - Dihidrolipoamida dehidrogenase
- Glutation reduktase
- Tioredoksin reduktase
|
|---|
| 1.8.2: sitokrom | - Sulfit dehidrogenase
- Tiosulfat dehidrogenase
- Flavositokrom c sulfida dehidrogenase
|
|---|
| 1.8.3: oksigen | |
|---|
| 1.8.4: disulfida | - Glutation-homosistina transhidrogenase
|
|---|
| 1.8.5: kuinon | - Glutation dehidrogenase (askorbat)
|
|---|
| 1.8.98: Lain-lain, diketahui | - Reduktase heterodisulfida CoB—CoM
|
|---|
| 1.8.99: Lain-lain | |
|---|
|
|---|
| 1.2.1: NAD atau NADP | - Aldehid dehidrogenase
- Asetaldehid dehidrogenase
- Dehidrogenase aldehid rantai panjang
- Formaldehid dehidrogenase bergantungan mikotiol
|
|---|
| 1.2.2: sitokrom | - Format dehidrogenase (sitokrom)
|
|---|
| 1.2.3: oksigen | |
|---|
| 1.2.4: disulfida | |
|---|
| 1.2.7: protein besi-sulfur | |
|---|
|
|---|
- Asetolaktat sintase
- Asil KoA dehidrogenase
- Faktor pencetus apoptosis
- Butiril KoA dehidrogenase
- Kriptokrom
- Sitokrom b5 reduktase
- Sitokinin dehidrogenase
- Dihidrolipoamida dehidrogenase
- Flavodoksin
- Metemoglobin reduktase
- Metilenatetrahidrofolat reduktase
- NADH dehidrogenase
- NADPH oksidase
- Nitrat reduktase
- Sarkosina oksidase
- Tioredoksin reduktase
|
|
|---|
| Fotosintesis | - Kompleks protein pusat tindak balas fotosintesis
- Fotosistem
|
|---|
| Dehidrogenase | |
|---|
| Lain-lain | - CAD
- Karbamoil fosfat sintase II
- Aspartat karbamoiltransferase
- Dihidroorotase
- Sistem berkandungan P450
- Kompleks sitokrom b6f
- Rantaian pengangkutan elektron
- Kompleks sintetase asid lemak
- Kompleks glisina dekarboksilase
- Protein trifungsi mitokondrion
- Sistem fosfotransferase gula fosfoenolpiruvat
- Poliketida sintase
- Kompleks sukrase-isomaltase
- Triptofan sintase
|
|---|
|
|---|
Glukosa-6-fosfat
isomerase Fruktosa-bisfosfat
aldolase 2 × 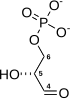 Gliseraldehid 3-fosfat
dihidrogenase 2 ×  2 × 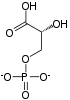 2 × 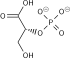 2 ×  2 ×  |

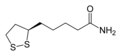 Lipoamida
Lipoamida Dihidrolipoamida
Dihidrolipoamida